En los laboratorios de investigación donde necesariamente se necesite obtener ADN purificado para la realización de los experimentos diarios o frecuentes, se utiliza gran cantidad de reactivos de la mejor calidad, por lo que resultan muy caros. En el caso de los llamados países del tercer mundo que no cuentan con suficientes recursos económicos, se hace necesaria la búsqueda de alternativas para la realización de estas investigaciones sin que por ello se afecte la calidad de los trabajos.
Por tal motivo realizamos un estudio aplicando las propiedades de la memoria del agua demostradas por el científico francés Jacques Benveniste y sustituimos algunas de las soluciones que se utilizan en el protocolo de purificación de ADN plasmídico, como la solución permeabilizadora de la membrana bacteriana, Sol I, (50mM Glucosa, 10 mM tris-HCl pH 8, 1mM EDTA), la solución que se utiliza en la ruptura o lisis de la membrana bacteriana, Sol II, (0.2 N NaOH, 1% SDS) y la solución que se utiliza para el precipitado de otras biomoléculas como proteínas, Sol III, (4M Acetato de potasio, 0.2 M Ácido acético) por las soluciones dinamizadas SolI 15CH, Sol II 15C y Sol III 15C respectivamente.
Obteniéndose ADN plasmídico purificado, siendo significativo los bajos rendimientos de ARN presentes después de la purificación usando las soluciones homeopatizadas, comparado con la cantidad de ARN que se obtiene con las soluciones tradicionales. Esto es muy importante, pues en muchos casos debemos eliminar el ARN presente después del proceso de purificación utilizando una enzima costosa (ARNasa).
El ADN obtenido en ambos fue digerido utilizando las enzimas HindIII y EcoRI y el patrón obtenido fue exactamente el mismo, lo que demuestra la efectividad del método.
Si las soluciones homeopatizadas son almacenadas correctamente pueden ser utilizadas durante 6 meses.
Introducción
En los laboratorios de Biología Molecular y Bioquímica es necesario el uso de ADN para realizar los diferentes experimentos que en ellos se realizan. Constantemente se diseñan y estudian nuevas construcciones para generar nuevos clones de bacterias y otros microorganismos. Por lo que se utilizan grandes cantidades de kits y soluciones, así como reactivos de la mejor calidad. Generalmente estas soluciones son costosas, sobre todo para los laboratorios que se encuentran en los países del llamado tercer mundo o subdesarrollados.
Los plásmidos son moléculas pequeñas de ADN circular capaces de replicarse de forma autónoma, independientemente del cromosoma. Son muy comunes en bacterias y algunos de ellos, los más pequeños, existen en las células bacterianas en un número elevado (hasta 100 copias por célula). Los plásmidos son una herramienta fundamental en Biología Molecular e Ingeniería Genética ya que se pueden usar como vectores para introducir en ellos cualquier fragmento de ADN de interés y de esta forma amplificarlo de forma natural dentro de la bacteria en la que el plásmido se replica (es lo que se llama clonar un fragmento de DNA).
Un método que permite visualizar ADN plasmídico o fragmentos del mismo es la electroforesis en gel de agarosa, en la cual se separan en función de su tamaño. Esta técnica consiste en someter la mezcla de moléculas de ADN embebidas en un gel de agarosa a un campo eléctrico. Las moléculas de ADN son atraídas al polo positivo debido a la carga neta negativa de ambas cadenas consecuencia de sus grupos fosfato. Las moléculas más pequeñas migran más rápido que las grandes al verse menos frenadas por el gel de agarosa en su desplazamiento. Cuando los fragmentos se
han separado, el gel se sumerge en una solución que contiene bromuro de etidio y luego se ilumina con luz UV.
El bromuro de etidio (EtBr) es una molécula plana con afinidad por el ADN que se intercala entre las pares de bases y que tiene la particularidad de que fluoresce cuando está unido a él. Al iluminar el gel con luz UV se ven bandas de fluorescencia que corresponden a moléculas de ADN.
Estudios realizados sobre la memoria del agua, donde se demuestra como aplicando el proceso de dilución homeopática a diferentes sustancias, diluciones superiores al número de Avogadro aun presentan actividad biológica y la información biomolecular aún se conserva exactamente igual, que la sustancia original;
Cuando utilizamos potencias mayores a la 12 centesimal, donde ya no existen moléculas de la sustancia de partida, es decir, una dinamización en la que la dilución es muy superior al número de Avogadro, se ha demostrado que la información energética de la tintura madre aún está presente y con mucha más energía [1,2].
De manera que desde una perspectiva física, la dinamización homeopática debe su actividad farmacológica al solvente modificado por el efecto de dilución sucusión. Para la física, el proceso de agitación transforma la organización molecular del solvente y le imprime una actividad específica haciendo que cada vez que se aumente en potencia la energía de la sustancia se incrementa.
Con el aumento del grado de potenciación, la sustancia activa pierde la masa molecular del soluto y el ámbito energético gana en importancia [1].
Las particulares características físicas del agua en especial su plasticidad y su variabilidad estructural dependiente de la temperatura permiten, por una parte, el almacenamiento de información y por otra, el transporte de esta información a todas las zonas del organismo. Esta transferencia de información desempeña una función central en el proceso de dinamización que se realiza en homeopatía [2].
En el presente trabajo se ha utilizado el método de preparación de los medicamentos que usa la homeopatía para la preparación de soluciones que se utilizan comúnmente en el proceso de purificación de ADN plasmídico. Para estos propósitos utilizamos la cepa BL21 [3] proveniente de la bacteria Escherichia coli, transformada con el plásmido pBR322[4]. Demostrando que al homeopatizar estas soluciones se obtiene ADN de igual calidad que el obtenido utilizando los métodos convencionales.
Materiales y Métodos
Protocolo para la purificación de ADN plasmídico.
1- Crecer un precultivo durante toda la noche (12h), en medio LB (Luria-Bertani) a la temperatura apropiada y antibiótico adecuado, con agitación.
2- Tomar 3ml del cultivo crecido, centrifugar a 16 000 x g 2 minutos.
3- Descartar el sobrenadante.
4- Resuspender el pellet en 1 ml de buffer STE, usando vortex.
5- Centrifugar a 16 000 x g 2 minutos.
6- Resuspender el pellet en 200?l de Sol I fría. Colocar los tubos en hielo.
7- Añadir 400 ?l de Sol II recién preparada, invertir suavemente e incubar en hielo 5 minutos.
8- Añadir 600 ?l de Sol III [Acetato de potasio (KAc 4/2)] fría. Agitar vigorosamente, dejar reposar en hielo 10 minutos.
9- Centrifugar a 16 000 x g 10 minutos.
10- Pasar 900?l del sobrenadante a otro tubo, con cuidado para no coger debris celular.
11- Añadir 450 ?l de Isopropanol, mezclar bien.
12- Centrifugar a 16 000 x g 5 minutos.
13- Decantar el sobrenadante, dar golpe de centrifuga (16 000 x g 15 segundos) para eliminar el sobrenadante residual.
14- Adicionar 500 ?l de etanol al 70 %, invertir los tubos y volver a centrifugar a 16 000 x g 5 minutos.
15- Eliminar el sobrenadante, dar golpe de centrifuga para eliminar el sobrenadante residual aspirando cuidadosamente con pipeta.
16- Secar ligeramente el pellet durante 5 minutos y resuspender en 35 ?l de TE 1x.
Buffer STE: 100mM NaCl, 10 mM Tris-HCl pH 8, 1 mM EDTA.
Solución I: 50mM Glucosa, 10 mM tris-HCl pH 8, 1mM EDTA.
Solución II: 0.2 N NaOH, 1% SDS.
Solución III: 4M Acetato de potasio, 0.2 M Ácido acético.
TE 1X: 10 mM Tris-HCl pH 8, 1mM EDTA.
Cepa utilizada: BL21 de Escherichia coli transformada con el plásmido pBR322.
Soluciones homeopatizadas
Fueron homeopatizadas las soluciones I (15C), II (15C) y Sol III (15C).
Se dinamizó agua estéril hasta la 15 C.
Se realizaron cuatro purificaciones de ADN plasmídico:
Purificación 1: se utilizó el protocolo sin ningún cambio
Purificación 2: se sustituyeron las soluciones I, y III por las dinamizaciones Sol I 15C y Sol III 15C.
Purificación 3: se sustituyeron las soluciones I, II y III por las dinamizaciones Sol I 15C, Sol II 15C y Sol III 15C.
Purificación 4: se sustituyeron las soluciones I, II y III por el agua estéril homeopatizada, vehículo usado para realizar las diluciones homeopáticas.
Control negativo: mezcla de las soluciones.
Visualización del ADN
Para visualizar el ADN se realizó un gel de agarosa al 1 %. En un matraz mezclar una cantidad adecuada de agarosa (1 g) con tampón TBE [Tris-borato-EDTA (etilen-diamino tetraacético)] (100 ml). La mezcla (agarosa + TBE) se calentó con la ayuda de un hasta que se fundió y la agarosa se disolvió completamente. Posteriormente se añadieron 5 ml de una solución de bromuro de etidio (10 mg/ml).
Se dejó enfriar ligeramente la mezcla (gel) hasta una temperatura de entre 50 ºC a 60 ºC y se vertió, sobre un molde o cama electroforética, previamente preparada, colocando el peine adecuado para formar los pocillos. Se dejó enfriar a temperatura ambiente hasta que gelificó completamente.
En un tubo eppendorf limpio se añadieron 20 ml de la muestra (10 ml del ADN purificado + 10 ml de agua) y 3 ml de tampón de carga.
La muestra se depositó en uno de los pocillos del gel de agarosa al 1% previamente fabricado usando una micropipeta (éste fue colocado con anterioridad sumergido en tampón TBE en una cubeta de electroforesis).
Se cerró la cubeta de electroforesis, se colocaron los electrodos de tal forma que el polo negativo quedara al lado de los pocillos y el polo positivo en el extremo opuesto del gel, hacia donde va a migrar el ADN y se aplicó una corriente eléctrica continua de 100-120 voltios.
La corrida de la electroforesis se detuvo después de 60 minutos. Se pasó a visualizar el ADN con ayuda de un transiluminador de luz ultravioleta de onda corta.
Digestión del ADN purificado
Para la digestión del ADN purificado se utilizaron las enzimas EcoRI y Hind III, las reacciones se realizaron como sigue: 1ug de ADN fue incubado con el buffer adecuado y 1u de enzima, las reacciones fueron realizadas a 37°C durante 1h.
Las enzimas y los buffer provienen de BioLabs.
Resultados y Discusión
La aplicación de las propiedades de la memoria del agua, pueden ser innumerables, nosotros nos dimos a la tarea de aplicar esta característica a soluciones usadas comúnmente en laboratorios de Biología Molecular e Ingeniería Genética para hacer un ahorro de reactivos y
evitar el contacto directo de los investigadores y técnicos con soluciones que pudieran resultar tóxicas para el organismo.
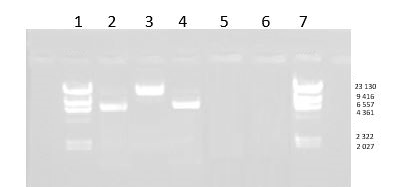
Se logró purificar ADN utilizando tanto las soluciones sin homeopatizar como utilizando las soluciones homeopatizadas, sin embargo cuando usamos agua homeopatizada no fue posible purificar ADN, tal y como se muestra en la figura 1.
Es significativo señalar que las soluciones fueron preparadas de forma tal que en todas se superara el límite de Avogadro (6.002045 x 10 23 moléculas por mol), por lo que en ellas no queda nada de la solución original.
Con la realización de este experimento demostramos que utilizando el método homeopático es posible transmitir información de una dinamización a otra, para así conservar las propiedades del soluto original, a partir del cual se realizaron las dinamizaciones.
Figura 1: Electroforesis en gel de agarosa 1%. Carriles 1 y 7 marcadores de peso molecular Lambda Hind III, carril 2: purificación 1, carril 3: purificación 2, carril 4: purificación 3, carril 5: purificación 4, carril 6: control negativo.
Podemos apreciar que cuando usamos las soluciones I y III se observa ADN pero al parecer es ADN cromosomal, este resultado nos sorprendió, pero al mismo tiempo lo consideramos de interés, ya que fue posible también obtener ADN cromosómico, utilizando soluciones homeopatizadas.
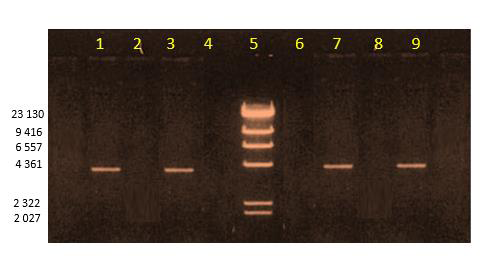
Figura 2: Electroforesis en gel de agarosa 1%. Digestión del ADN purificado utilizando las enzimas de restricción EcoRI y Hind III. 1-4 (EcoRI). 6-9 (HindIII). 1,7(purificación 1), 2,8 (purificación 2); 3,9 (purificación 3); 4,6 (purificación 4).
En la figura 2 podemos apreciar la digestión realizada al ADN obtenido, como se puede ver no hay diferencias entre el ADN obtenido con las soluciones convencionales y las soluciones homeopatizadas. El tamaño de la banda obtenida después de la digestión corresponde con el tamaño esperado de 4361 pb.
Este método está sujeto a cambios ya que se requiere probar dinamizaciones más altas y combinarlas para lograr optimizar completamente el proceso, pero lo más importante es el resultado significativo de la obtención de ADN utilizando soluciones dinamizadas, lo cual sugiere que la homeopatía puede tener otras aplicaciones adicionales a las que ya se conocen.
Este método resulta muy ventajoso y ecológico, ya que nos permite el ahorro de reactivos y nos evita la manipulación de sustancias que podrían llegar a ser perjudiciales para el organismo humano.
Bibliografía
1- Benveniste J. Highly dilute antigen increases coronary flow of isolated heart from immunized guinea pigs. FASEB J. 1992;6
2- Schulte J, Endler PC. Update on preliminary elements of a theory of ultra high dilutions. Homeopathy. 2015 Oct;104(4):337-42.
3- Amersham Biosciencies.2002. GST Gene fusion System Handbook. USA.
4- Watson, N. (1988). «A new revision of the sequence of plasmid pBR322». Gene 70 (2): 399403.
Dra. Niurka Meneses Moreno
Universidad de Berna, Suiza
Kevin, nuestro redactor oficial en Farmacia Coliseum, tiene más de 10 años de experiencia en el sector farmacéutico. Con un enfoque claro y accesible, convierte temas complejos en consejos prácticos y útiles, ayudando a nuestra comunidad a mejorar su salud y bienestar.